BioEdit یک ویرایشگر توالی نوکلئوتیدی رایگان در زیست شناسی مولکولی
نرم افزار BioEdit یک ویرایشگر توالی نوکلئوتیدی رایگان و یکی از رایج ترین نرم افزارهای مهم مورد استفاده در مطالعات زیست شناسی مولکولی است که در 20 فوریه 2009 منتشر شد.
نرم افزار BioEdit یک ویرایشگر توالی نوکلئوتیدی رایگان است که موارد استفاده آن برای ویرایش توالی های زیستی و نتایج حاصل از مقایسه یا الاینمنت توالی ها است و توسط یک دانشجوی فارغ التحصیل علوم زیستی و به جهت افزایش سهولت ویرایش توالی ها نوشته شده است. نویسنده نرم افزار BioEdit آن را یک رابط بصری مستند چندگانه با ویژگی های مناسب می نامد.
این نرم افزار امکان ویرایش توالی ها و برخی ویژگی های پایه و همچنین برخی امکانات ساده آنالیزی را به صورت رایگان ارائه می دهد. اما با نگاهی دقیق تر به این نرم افزار متوجه خواهید شد که BioEdit یک نرم افزار توانمند است که می تواند امکانات بی شماری به کاربران ارائه دهد.
باز کردن فرمت های مختلف توالی های زیستی از جمله فرمت های استاندارد بانک ژن مانند GB، GEN، GNK ، GBK و فرمت های استاندارد FASTA، فرمت های الاینمنت PIR و NBR، فرمت های Clustal ، phylip و بسیاری از فایل های مرتبط با توالی و الاینمنت توسط این نرم افزار امکان پذیر است و به عنوان یک ابزار سریع و ساده وجود آن بر روی کامپیوتر های شخصی بسیار مفید خواهد بود.
هر توالی که مورد نیاز شماست توسط این نرم افزار باز خواهد شد و هیچ فرمت نا آشنای مربوط به علوم زیستی بر روی کامپیوتر شخصی خود نخواهید داشت. جالب است بدانید، مجوز نرم افزار رایگان و ماژول های به روز آن در کنار توانایی سریع آن در تولید نتایج، آن را به یکی از محبوب ترین برنامه های زیست شناسی مولکولی امروزه تبدیل کرده است.
نرم افزار BioEdit را می توانید به منظور ساخت قطعات مکمل از روی توالی خود، آنالیز مشخصات پرایمرها، بررسی نقاط برش آنزیمهای برش دهنده و مقایسه توالی ها استفاده کرد. نکته مهم در این نرم افزار این است که در بخش توالیهای پروتئینی هم این نرم افزار بسیار کامل و مفید خواهد بود.
امکانات این نرم افزار بسیار گسترده است. بیو ادیت با تمام پایگاه های داده توالیهای زیستی امکان ارتباط دارد و با توجه به اینکه از آن دسته از نرم افزارهای رایگان است، کاربر در زمان استفاده مشکلی از بابت لایسنس نرم افزار نخواهد داشت.
پروتکل ها/دستورالعمل های استفاده از BioEdit / Mega2
1) برای باز کردن توالی ها در نرم افزار BioEdit:
الف) در بخش منو گزینه فایل و سپس گزینه Open را انتخاب کنید تا اولین توالی شما را باز کند.
ب) در منوی File گزینه Import را انتخاب کنید، احتمالاً مجبور خواهید بود که آنرا تغییر دهید.
سپس در بخش files of type ،فقط گزینه all files را انتخاب کنید.
با نگه داشتن دکمه control و کلیک بر روی آن، فایل بعدی یا همه آنها را انتخاب کرده و سپس Enter را بزنید.
2) زمانی که تمام توالیها در جای خود قرار گرفتند، باید آنها را تراز (در یک ردیف قرار دهید) کنید. برای انجام این کار :
الف) عناوین توالی را در سمت چپ صفحه نمایش خود هایلایت کنید و در منوی بالای صفحه گزینه Edit را انتخاب کرده و سپس در پنجره باز شده گزینه Copy Sequence(s) را کلیک و توالی(ها) را کپی کنید.
ب) به آدرس http://www.ebi.ac.uk/clustalw/ کلیک راست کنید و در پنجره باز شده گزینه Paste را انتخاب کنید. سپس دکمه Run را بزنید.
ج) در نهایت شما یک صفحه نمایش نتایج دریافت خواهید کرد. دقیقاً بالاتر از توالی های تراز شده ی شما عددی خواهد بود که متعاقب آن است. روی (aln)کلیک راست کرده و گزینه Save Target As را کلیک کنید (این را به عنوان یک فایل متنی ذخیره نکنید، زیرا برنامه بعدی که استفاده خواهید کرد قادر به خواندن آن نیست!)
د) سپس به نرم افزار BioEdit بازگردید با استفاده از فایلی که اخیراً از آن ذخیره کرده اید، و فایل جدیدی را باز کنید.
ه) در قسمت بالای توالی یک نوار منوی وجود دارد که تعداد زیادی چیز جالب در آن قرار دارد. بررسی کنید که چه کارهایی می شود انجام داد. حروف رنگی کدهای اسیدهای آمینه را رنگ میکنند و آمینواسیدها و حروف رنگی با نقاط جدا میشوند اسیدهای آمینه در همه توالی ها یکسان هستند به این ترتیب شما فقط تفاوت ها را می بینید.
- فایل خود را به عنوان یک فایل fasta ذخیره کنید. اگر aln باقی مانده است آن را قبل از ذخیره حذف کنید.
3) برنامه Mega2 را باز کنید.
الف) به شما سه گزینه ارائه خواهد شد:
- Click me to activate a data file
- Go to the MEGA2 web page
- Citing MEGA2 in publications
ب) فایل aln را که قبلاً از اینترنت یا فایل fasta ذخیره کرده اید، باز کنید.
پیام ذخیره شده از BioEdit که به احتمال زیاد فقط پیام error expected است را دریافت خواهید کرد سپس گزینه OK را کلیک کنید.
ج) باید هر فایل را به یک فایل با فرمت مگا(Mega) تبدیل کنید. برای انجام این کار منوی Utilities را انتخاب کرده و Convert to Mega format را انتخاب کنید. این پرونده جدید را ذخیره نمائید. این یک اسم کمی متفاوت است تا بتوانید راحت تر آن را پیدا کنید. اکنون این فایل را ببندید.
د) برای فعال کردن دوباره یک فایل داده (data file) کلیک کنید و فایلی را که تازه ذخیره کرده اید انتخاب کنید و بیان کنید که این یک توالی نوکلئوتیدی و صحیح است.
ه)از شما سؤال خواهد کرد که آیا توالی نوکلئوتیدی کد کننده پروتئین دارید؟
اگر cyt b را کلیک کنید به معنی بله اگر حلقه D (D-loop click) را کلیک کنید یعنی نه. (اگر بله را انتخاب کردید، مجبورید به انتخاب میتوکندری مهره داران هستید.)
و) در فهرست منو، گزینه Data Explorer را انتخاب کنید و نگاهی به توالیهای خود بیندازید.
-در منوی پایین بر روی حروف روشن/ قرمز رنگ کلیک کنید.
توالی ها را ببینید که چگونه آنها تغییر می کنند:
C= نوکلئوتیدها
V=متغیر
– در منوی بالای صفحه گزینه phylogen را انتخاب کرده سپس یک پنجره باز می شود و گزینه UPGMA را انتخاب کنید.
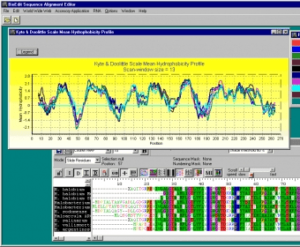
-در زیر تنظیمات آنالیز گزینهModel’ (منوی کشویی) را با عنوان: (Nucleotide > No. of Differences)تنظیم کنید سپس گزینه ok را انتخاب نمائید، که یک درخت ایجاد میشود، شما باید پنجره ای را انتخاب کنید که Tree نام دارد. در بالای منوهایی با چندین نوع درخت شاخه بر روی آن مشاهده خواهید کرد. همه آنها را امتحان کنید.
این روش های مختلفی برای نمایش داده های شما و روابط آن ها هستند. روشی متفاوت را امتحان کنید. در زیر منوی فیلوژنی، Neighbor joining را انتخاب کنید با استفاده از همان مدل مشابه در بالا بپیوندید. هر دو درخت را باز کنید و با استفاده از همان سبک خروجی آنها را با هم مقایسه می کند. آیا تفاوت قابل توجهی با هم دارند یا خیر.
بیشتر بخوانید:

برای آموزش کار با نرم افزار BioEdit و سایر نرم افزارهای آزمایشگاهی به صورت خصوصی و گروهی
از طریق لینک دوره های آموزشی تخصصی آزمایشگاه می توانید اقدام نمایید











































سلام من چندتا سوال داشتم میشه راهنمایی کنید
با سلام
تمامی سوالات خود را زیر پست های مربوط به آنها می توانید مطرح نمایید، کارشناسان ما در سریع ترین زمان ممکن پاسخگوی شما خواهند بود.
وقت بخیر
علت ارور ۰۱۳ در بایو ادیت چیست؟باتشکر