توالی یابی SOLiD یا Sequencing by Oligonucleotide Ligation and Detection یکی از روش های تعیین توالی نسل دوم
تکنولوژی(Sequencing by Oligonucleotide Ligation and Detection) SOLiD از روش های نسل دوم توالی یابی است که در سال 2006 ارائه گردید و در اواخر سال 2007 توسط شرکت ABI (Applied Bioscience) خریداری و تجاری سازی شد. از این فناوری برای توالییابی DNA استفاده میشود، اما به دلیل ماهیت موازی با توالی یابی نسل جدید، آنها در ترنسکریپتومیکس و اپی ژنومیکس نیز کاربرد دارند.
روش میکرواری زمانی پایه اصلی ترنسکریپتوم در گذشته بود و فناوری های مبتنی بر میکرواری متعاقباً به حوزه های دیگر منشعب شد. با این حال، محدودیتی که وجود دارد این است که فقط می توان اطلاعاتی را برای کاوشگرهایی که روی تراشه هستند به دست آورد. رونویسی RNA-Seq با توالی یابی نسل جدید به این معنی است که این موانع دیگر درست نیستند. کل رونوشت هر ارگانیسم میتواند به طور بالقوه در یک اجرا (برای ژنومهای باکتریایی بسیار کوچک) توالییابی شود و نه تنها شناسایی هر رونوشت در دسترس است، بلکه دستیابی به پروفایل بیان نیز ممکن است، زیرا میتوان به خوانش های کمی نیز دست یافت.
CHIP-Seq نیز روشی برای تعیین مکان های اتصال فاکتور رونویسی و برهمکنش های DNA-پروتئین است که توالی یابی نسل جدید نیز می تواند در این زمینه اعمال شود.
در روش توالی یابی SOLiD هر نوکلئوتید موجود در هر یک از قطعات 2 مرتبه، در 2 واکنش اتصال مستقل شناسایی می شود و در نتیجه مهم ترین مزیت این روش تعیین توالی میزان صحت %99.94 آن می باشد، از این رو، دقت توالی یابی در این روش در مقایسه با روش های دیگر در سطح بالایی است. همچنین سیستم این فناوری بر خلاف سیستم Roche 454 FLX توسط هموپلیمرها مانع نمی شود و بنابراین مناطق تکراری هموپلیمر بزرگ و دشوار دیگر برای توالی یابی مشکلی ندارند.
مهم ترین محدودیت این روش کوتاه بودن طول توالی خوانده شده می باشد که 35 تا 75 نوکلئوتید است. در ابتدا، در هر ران دستگاه تا 3GB توالی خوانده می شد، اما در دسامبر 2009، نسخه ی جدیدی از این تکنولوژی با نام Plus 3TM SOLiD معرفی گردید که می تواند در هر اجرا، بیش از 60GB توالی را بخواند.

در روش SOLiD همانند روش پایروسکئونسینگ (Pyrosequencing) در ابتدا کتابخانه DNA تهیه شده و سپس تکثیر قطعات DNA بر روی دانه های پوشیده از آغازگر که مهره های پارامغناطیسی 1μΜ هستند با روش emPCR (emulsion PCR) انجام می گیرد. سپس هر یک از این مهره ها روی سطح شیشه ای (SOLiD flow well) به طور تصادفی توزیع شده و تعیین توالی به وسیله آنزیم لیگاز به جای پلیمراز (Sequencing by Ligation) صورت می گیرد.
برای این کار ابتدا آغازگر عمومی به یکی از سازگار دهنده ها جفت شده و پس از آن کاوشگرهای فلورسنت 8 نوکلئوتیدی به منظور جفت شدن با قطعات حاصل از تکثیر در مجاورت آغازگر عمومی با یکدیگر رقابت می کنند. کاوشگرهای الیگو نوکلئوتیدی دارای جایگاه اتصال در اولین باز، جایگاه برش در پنجمین باز و 4 رنگ فلورسنت مختلف متصل به آخرین باز می باشند. از آنجا که 2 باز ابتدایی تعیین کننده نوع رنگ فلورسنت متصل به کاوشگر بوده و به طور هم زمان 2 نوکلئوتید را بررسی می کنند به کاوشگرها di-base Probes گفته می شود.
با جفت شدن کاوشگرهای الیگو نوکلئوتیدی به رشته الگو سیگنال فلورسنت ثبت می شود و به وسیله ی برش شیمیایی بین پنجمین و ششمین نوکلئوتید کاوشگر سیگنال حذف می شود. پس از برش، کاوشگر اکتامر دیگری متصل شده و چرخه اتصال جدیدی شکل می گیرد.
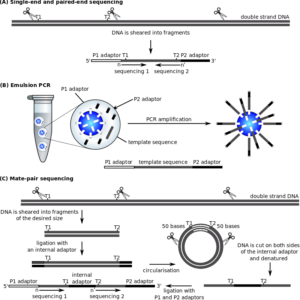
با این روند از گروه پنج تایی 2 باز ابتدایی آن شناسایی می شود و سرانجام تمام قطعه به صورت دو رشته ای در می آید. سپس رشته تازه سنتز شده و آغازگر عمومی از رشته DNA اولیه جدا می گردد و آغازگر عمومی جدیدی که به اندازه یک نوکلئوتید از آغازگر قبلی کوتاه تر است (n_1) اضافه می گردد. به منظور تعیین توالی تمامی بازها این فرایند پنج بار و با پنج آغازگر عمومی با اندازه n تا n_4 تکرار می شود.








































