انواع روش های توالی یابی از ابتدا تاکنون
مروری بر روش های توالی یابی از سنگر تاکنون
روش ها و تکنیک هایی که امروزه برای توالی یابی مورد استفاده قرار می گیرند به دو دسته تقسیم می شوند:
- روش خاتمه زنجیر اتومات سنگر
- توالی یابی نسل جدید شامل مجموعه ای از روش های مختلف با امکان تعیین میلیون ها توالی به طور همزمان.
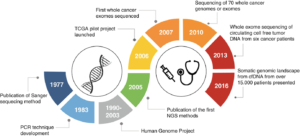
12 سال پس از انتشار ساختار DNA دو رشته ای توسط واتسون و کریک در سال 1953، اولین مورد توالی یابی پلی نوکلئوتیدی طبیعی (tRNA) گزارش شد. در آن زمان دانشمندان تنها قادر به توالی یابی تعداد کمی باز(نوکلئوتید) در سال بودند و این میزان برای توالی یابی کل ژنوم کافی نبود. نزدیک به 10 سال طول کشید که موفقیت های چشمگیری در زمینه توالی یابی به دست آید و روش هایی همچون سنگر و ماکسام گیلبرت ابداع شدند. همزمان بودن این اتفاق با ابداع روش PCR و کشف آنزیم Reverse Transcriptase کمک شایانی به توالی یابی RNA و همچنین بررسی قطعات تکثیرشده کرد.
توالی یابی ماکسام گیلبرت نیز عاری از عیب نبود و از معایب آن می توان به استفاده از مواد شیمیایی خطرناک، پیچیده بودن مراحل آماده سازی و عدم استفاده از آن برای ژنوم های طولانی همانند ژنوم انسان اشاره کرد. همچنین از مهم ترین معایب تعیین توالی به روش سنگر، استفاده از ژل الکتروفورز می باشد. در روش کاپیلری نیز که توان تعیین توالی 96 نمونه در یک زمان را داراست، محدود به kb 30-60 در 3-4 ساعت جریان الکتروفورز است.
با پیشرفت علم همزمان ده ها روش جدید برای توالی یابی ارائه شد اما در این میان روش سنگر اتومات شده روش غالب بود. علی رقم دقت بالا این روش بسیار پرهزینه بود و در مقیاس بزرگ عملا کارایی نداشت و نیاز به Assemble کردن توالی ها بود.
در این زمان همچنین شاهد ظهور تکنولوژی Microarray در زمینه مطالعات بیان ژن و همچنین بررسی تغییرات نوکلئوتیدی بودیم اما همچنان ضعف در سیستم توالی یابی وجود داشت و از این رو روش های جدید با عنوان روش های نسل جدید توالی یابی یا به اختصار NGS پا به عرصه ظهور گذاشتند.
به دلایلی که گفته شد، در سال 2000 با پیشرفتهای ایجاد شده در زمینههای نانوتکنولوژی و بیوانفورماتیک روشهای جایگزینی به منظور افزایش سرعت و افزایش بازدهی تعیین توالی DNA ابداع شد که اصطلاحا Next Generation sequencing (NGS) نامیده می شوند و نیازی به ژل الکتروفورز که فرایندی طولانی و زمان بر است ندارند و دارای تفاوت چشمگیری با تکنیکهای قبلی بودند، یکی از موارد شاخص این روش ها، تعیین توالی با سنتز (sequencing by synthesis) می باشد. به زبان دیگر، توانایی مانیتور کردن هم زمان نوکلئوتیدهایی که به رشته در حال تکثیر DNA افزوده می شوند وجود دارد.
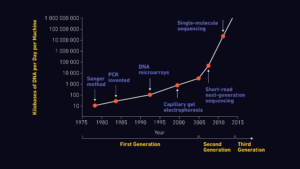
روش های نسل سوم توالی یابی نیز که هم اکنون بیشتر در حال طی مراحل آزمایشی است براساس سرعت بالاتر و کاهش هزینه ها در حال توسعه است. در این روش ها برخلاف روش های نسل دوم دیگر نیازی به مرحله تقویت یا تکثیر توالی ها برای ساخت کتابخانه نیست و اساس آن بر توالی یابی مولکول به شکل تک(یکجا) است.
اهمیت و کاربردهای توالی یابی نسل جدید
تقریبا تمام صفات و اطلاعات وراثتی بر اساس ساختار عملکردی ژنوم ارگانیسم است از اینرو اهمیت NGS به منظور درک و بررسی این مکانیسم ها جهت دستیابی به اهداف بهتر حائز اهمیت است. علاوه بر حوزه ژنومیک، NGS در سایر حوزه های مختلف از جمله مطالعات بیان ژنی، مطالعات اپی ژنتیک و حوزه متاژنومیکس کارایی و عملکرد مناسب داشته است که در ادامه راجع به آنها بحث خواهد شد.
-
ژنومیکس
1- توالی یابی کل ژنوم(WGS)
شناسایی واریانتها با استفاده از توالی یابی کل ژنوم انجام می شود. توالی یابی کل ژنوم یا به اختصار WGS تاثیر بسزایی در مطالعه باکتری ها، ویروس ها، قارچ ها و همچنین حوزه مطالعات پستانداران از جمله انسان داشته است. تکنیک WGS کمک شایانی به شناسایی ارتباطات تغییرات ژنومی مرتبط با بیماری ها کرده است. سیستم های جدید و پیشرفته NGS همچنین کمک فراوانی به دانشمندان در مطالعه گونه های جدید بیماری زا نموده است.
2- توالی یابی اگزوم(WES)
توالی یابی کل اگزوم یا به اختصار WES رایج ترین روش توالی یابی هدفمند است. این روش کمتر از 2 درصد ژنوم انسان را در برمی گیرد اما به دلیل اینکه بیشتر واریانت های بیماری زا در ناحیه کد کننده پروتئینی یا به اصطلاح اگزونی قرار دارند این روش نسبت به WGS بسیار به صرفه تر است. در واقع در این روش بخش کد کننده پروتئین های ژنوم انسان به شکل هدفمند انتخاب شده و مورد توالی یابی قرار می گیرد.
3- توالی یابی نقاط هدف (Targeted Sequencing Panel)
نوع دیگری از NGS است که در آن گروهی از ژن ها یا یک ناحیه خاص ژنومی جداشده و توالی یابی می شوند. برای این روش طیف گسترده ای از کیت های توالی یابی موجود است که تمرکز آنها بر ناحیه خاصی است، برای مثال ژن های دخیل در سرطان، اوتیسم یا بیماری های قلبی. این تکنیک در مقایسه با WGS و WES از میزان پوشش بهتر و بالاتری برخوردار است از این رو توانایی یافتن واریانت های بیماری زا را با دقت بیشتری دارد.
4- توالی یابی ژنوم جدید (de novo sequencing)
متدی از توالی یابی سطح ژنوم است که بدون در اختیار داشتن ژنوم رفرنس انجام می پذیرد.
-
ترانسکریپتومیکس
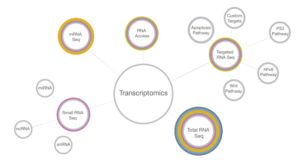
توالی یابی ترانسکریپتوم پیشرفت بزرگی در زمینه مطالعات بیان ژنی به ارمغان آورده، از این پس دیگر نیازی به مطالعات محدود یا گروه خاصی از ژن ها نیست و کل محتوی ترانسکریپتوم می تواند به شکل یکجا مورد بررسی قرار گیرد. در واقع با استفاده از این تکنیک می توان یک دید بیانی جامع از فرآیندهای داخل سلولی ارائه کرد. از جمله توانایی های دیگر آن می توان به شناسایی ایزوفرم ها، ترانسکریپت های جدید و شناسایی الحاق های ژنی(gene fusion) اشاره نمود.
1- توالی یابی نقاط هدف RNAها / Targeted RNA-seq
این روش جهت بررسی RNA های گروه های خاص بیانی مورد استفاده قرار می گیرد برای مثال کیت های هدفمند TruSeq شامل پنل هایی است که برای بررسی مسیرهای خاص مانند آپوپتوز، مسیر NFKB و سایر مسیرهای پراهمیت توسعه یافته است.
2- Small RNA and Non-Coding RNA-Seq
RNA های کوچک غیرکدکننده یا microRNA ها توالی نوکلئوتیدی بین 18-22 بازی دارند که نقش آنها تنظیمی است و معمولا سبب القا یا سرکوب بیان ژن می شوند. مطالعه microRNA در دهه اخیر سبب درک بهتر فرآیند بیان ژن شده است. در شکل زیر حوزه مطالعات ترانسکریپتوم آورده شده است.
-
اپی ژنومیکس
در حالی که حوزه ژنومیک درگیر بررسی تغییرات وراثتی یا اکتسابی توالی DNA است اپی ژنتیک (Epigenomics-Seq) تغییرات فعالیت ژن ها که به وسیله مکانیسم هایی غیر از تغییرات توالی هستند را بررسی می کند. مکانیسم های اپی ژنتیک شامل متیلاسیون DNA ، برهمکنش های DNA-پروتئین و تغییرات هیستونی است.
1- توالی یابی متیلاسون / Methylation Sequencing
یکی از موضوعات مهم در حوزه اپی ژنتیک مطالعه متیلاسیون سیتوزین در سراسر نواحی تنظیمی شامل پروموتر و هتروکروماتین است. این تغییر می تواند سبب بازآرایی کروماتین و القا بیان ژن شود. در حالی که روش های زیادی برای مطالعه اپی ژنوم وجود دارد توالی یابی نواحی متیله شده توسط تکنیک NGS یک دید جامع ژنومی از این تغییرات به ما می دهد. NGS برای مطالعه این نواحی از دو روش WGBS و RRBS بهره می برد که روش اول برای مطالعه کل ژنوم کاربردی است.
2- توالی یابی کروماتین ایمونوپرسیپیتاسیون / Chip-Seq
برهمکنش DNA-پروتئین و RNA-پروتئین تاثیر مهمی بر فرآیندهای داخل سلولی و شرایط بیماری زایی دارد این برهمکنش ها می تواند بوسیله NGS و با کمک تکنیک Chromatin Immunoprecipitation مورد بررسی جامع قرار گیرد. پروتکل های به کار رفته در این روش جهت مطالعه پروتئین های TF و سایر پروتئین های متصل شونده به DNA یا RNA مناسب است.
3- توالی یابی قطعات mRNA داخل ریبوزوم / Ribosome Profiling
یک روش برای توالی یابی قطعات mRNA داخل ریبوزوم هاست. خالص سازی و توالی یابی این قطعات یک دید کلی از فعالیت ریبوزوم ها در یک بازه زمانی خاص ارائه می دهد. این اطلاعات می تواند نشان دهد که چه پروتئین هایی داخل سلول به شکل فعال بیان شده اند. در واقع با استفاده از NGS یک آنالیز دقیق از پروفایل بیانی لحظه ای پروتئین می توان ارائه نمود.

برای کسب اطلاعات بیشتر و شرکت در دوره آنالیز داده های NGS (توالی یابی نسل جدید)
نسل اول توالی یابی
-
توالی یابی سنگر
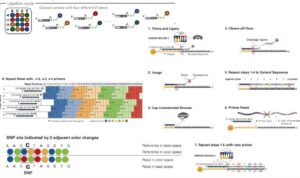
توالی یابی سنگر(Sanger) به نحوی زیربنای همه تکنولوژيهای توالی یابی است که هم اکنون مورد استفاده قرار میگیرند و ژنومیکس (علم مطالعه ژنوم) را شکل داده است. جهت تعیین توالی ژنومهای فراوانی مانند E.coli، مگس سرکه، کرمهای نواری (نماتودها)، موش و انسان از تکنیک توالی یابی sanger استفاده شده است.
در تکنیک توالی یابی سنگر (Sanger) مولکول های DNA تک رشته ای که فقط در حد یک نوکلئوتید با هم تفاوت دارند، می توانند توسط الکتروفورز در ژل پلی آکریل آمید حاوی اوره به عنوان ترکیب دناتوره کننده و یا سایر ترکیبات دناتوره کننده که باعث می شود DNA به صورت تک رشته ای باقی بماند با ولتاژ بالا از یکدیگر جدا شوند.
توالی یابی سنگر (Sanger) مثل فرایند تکثیر DNA و تکنیک PCR به پرایمر، DNA پلیمراز، توالی الگوی تکرشتهای و دئوکسی نوکلئوتیدها نیاز دارد. در ابتدا سوبسترای واکنش عمدتا DNA نوترکیبی بود که به وسیله دناتوراسیون می توانست به پرایمر توالییابی اختصاصی رشته متصل شود. قطعات DNA در وکتورهای فاژمیدی هم چون وکتور M13 که در اثر دستکاری میتوانستند DNA های نوترکیب تکرشتهای تولید کنند، کلون میشدند.
از اتورادیوگرافی جهت رؤیت نتیجه استفاده می کنند. خواندن اتورادیوگرام از سمت پایین ژل به بالا صورت می گیرد و در این حالت توالی های به دست آمده مکمل توالی اصلی می باشند. داده ها نیز به صورت دستی وارد کامپیوتر میشوند. به همین دلیل این پروسه بسیار طولانی است، ۱۲ ساعت برای الکتروفورز، ۱۲ ساعت برای ایجاد اتورادیوگرام و مدت زمان بسیار طولانی تری برای خواندن توالیها موردنیاز است.
همچنین احتمال وقوع اشتباه نیز بالا و استفاده از بازهای لیبل شده با مواد رادیواکتیو خطرناک است به همین دلیل از تعیین توالی سنگر به روش اتومات استفاده می شود که کارآیی آن را افزایش می دهد. در این روش توالی های تا 400 نوکلئوتید را می توان تعیین کرد.
بیشتر بخوانید: توالی یابی سنگر (sanger)، نسل اول تعیین توالی DNA
-
تعیین توالی سنگر به روش اتومات
با توجه به آن چه گفته شد انجام برخی اصلاحات الزامی بودند به خصوص اگر قصد ما از توالی یابی، ژنوم های بزرگی مثل ژنوم انسان بود. به همین منظور در اوایل دهه 1990 ماشین های توالی یابی اتوماتیک به صورت تجاری به بازار عرضه شدند که هم سریع تر بودند و هم احتمال خطا در آن ها به مراتب کم تر شد. اگر چه هزینه راه اندازی این تکنیک به دلیل گران بودن دستگاه آنالیز کننده توالی بسیار بالاست ولی از آنجایی که آنالیز چندین نمونه به صورت هم زمان است در نهایت هزینه توالی یابی هر نمونه بسیار پایین است.
مراحل انجام واکنش دقیقا همانند PCR معمولی است که تکثیر در دستگاه Thermal cycler انجام می گیرد. در هنگام پلیمریزاسیون، این امکان وجود دارد که دی دئوکسی نوکلئوتیدها متصل شده و زنجیره DNA خاتمه یابد. نسبت دی دئوکسی نوکلئوتیدها به دئوکسی نوکلئوتیدها باید طوری باشد که توقف همانندسازی حتما در هر کدام از بازهای A، G، T و C صورت بگیرد.
-
توالی یابی ماکسام گیلبرت
تکنیک ماکسام-گیلبرت که از روش های نسل اول توالی یابی است در سال ۱۹۷۶–۱۹۷۷ توسط الن مکسام و والتر گیلبرت جهت تعیین توالی DNA ابداع گردید. این روش بر اساس هضم شیمیایی و بدون نیاز به پلیمریزاسیون قطعه مورد نظر انجام می شود.
این تکنیک 2 سال پس از روش توالی یابی سنگر معرفی گردید و به سرعت محبوب تر شد زیرا در این تکنیک بر خلاف روش سنگر، به وکتور DNA تک رشته ای و پرایمر نیاز نبود و DNA تخلیص شده می توانست به طور مستقیم استفاده گردد. به این روش، روش تخریب شیمیایی یا روش End-Labling می گویند که با استفاده از آنزیم عملی می شود.
بیشتر بخوانید: مراحل انجام توالی یابی ماکسام گیلبرت
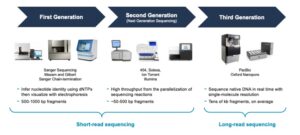
توالی یابی نسل جدید
نسل دوم و سوم روش های تعیین توالی به عنوان نسل جدید شناخته شده هستند. تفاوت اصلی این تکنیک ها به خاطر طراحی متفاوت آنها است که همزمان قادر است حجم بالایی از داده ها از نمونه های مختلف در مقیاس بزرگتر و پوشش با درجه بالاتر را تولید کند. این روش ها همچنین نسبت به حجم تولیدی داده ها بسیار مقرون به صرفه هستند. نقطه قوت NGS که سبب این توانایی ها شده، تغییر در نحوه آماده سازی کتابخانه های DNA/RNA و فرآیند توالی یابی آنهاست که شامل مراحل شکستن قطعات، اضافه کردن Adapter و همچنین تقویت قطعات است.
نسل دوم توالی یابی
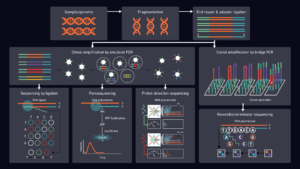
توالی یابی نسل دوم با استفاده از روش سنتز و یا روش لیگیشن انجام می شوند
بر اساس سنتز:
- Pyrosequencing
- Reversible Terminator
- Detecting of Hydrogen
بر اساس لیگیشن
- Hybridization and Ligation
-
توالی یابی پایروسیکوئنسینگ
نخستین روش توالی یابی بر اساس سنتز با امکان مانیتور کردن همزمان نوکلئوتیدهایی که به رشته در حال تکثیر DNA افزوده می شوند. توالی یابی به روش سنگر، اتوماتیک و کاپیلری و ماکسام گیلبرت همگی از توالی یابی های نسل اول هستند. این روش توسط مصطفی رونقی و پال نیرن در سال 1996 ابداع شد که در آن اندازه گیری پیروفسفات آزاد شده و دریافت سیگنال در حین سنتز مورد توجه است. این تکنولوژی توسط کمپانی Roche خریداری و به تولید رسید و با نام تجاری Roche 454 Sequencing به بازار عرضه شد.
پیروسکانس برای بررسی پلی مورفیسم های تک نوکلئوتیدی(SNP) به کار گرفته شد. البته علی رغم تفاوتها در روش سنگر و پیروسکانس، اساس توالی یابی در هر دو روش سنتز رشته اسید نوکلئیک است که تکنیک SBS نامیده میشوند به دلیل اینکه هر دو نیاز به عمل مستقیم DNA پلیمراز برای تولید و مشاهده محصول دارند. تفاوت عمده این روش با روشهای دیگر به کارگیری از dNTP های برچسب دار رادیواکتیو یا فلئورسنت بود. در واقع در این روش از لومینسانس برای اندازگیری سنتز پیروفسفات استفاده می شود.
در این روش DNA پلیمراز با کمک dNTP و الگوی تک رشته به سنتز DNA می پردازد، اما به جای استفاده هم زمان از هر چهار نوع dNTP، نوکلئوتیدها را به صورت مجزا به محیط واکنش می افزایند، هنگامی که dNTP درست افزوده شود به صورت dNMP به رشته در حال سنتز ادغام می گردد و گروه پیروفسفات آزاد می شود، اما اگر dNTP نادرست باشد به وسیله ی آنزیمی به نام آپیراز (Apyrase) که یک آنزیم نوکلئوتیداز می باشد، حذف می گردد و هیچ نوری ساطع نمی شود.
قرار گرفتن باز درست در دومین مرحله واکنش با بررسی نور ساطع شده مانیتور می گردد؛ (PPi) پیروفسفات آزاد شده توسط آنزیم ATP سولفوریلاز به ATP تبدیل می شود. ATP در واکنش دیگری توسط آنزیم لوسیفراز که لوسیفرین را به اکسی لوسیفرین تبدیل می کند باعث ساطع شدن نور می گردد و در نهایت این سیگنال نوری توسط دوربینهای حساس به سیگنالهای ساطع شده موسوم به CCD به صورت یک پیک ثبت و اندازه گیری میشود.
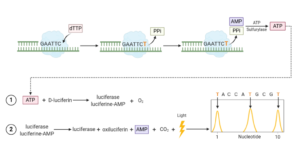
در مورد توالی یابی به روش پایروسیکوئنسینگ(Pyrosequencing) بیشتر بخوانید.
-
توالی یابی ایلومینا
پس از اولین روش تعیین توالی نسل دوم تحت عنوان پایروسیکوئنسیگ ،شرکت Solexa در سال 2006 دستگاه تعیین توالی خود تحت عنوان Genome Analyzer II را معرفی کرد که سال بعد امتیاز آن توسط شرکت Illumina خریداری شد. در این روش که نوعی تعیین توالی بر اساس سنتز می باشد، ابتدا آغازگرهای الیگو نوکلئوتیدی به صورت متراکم از طریق پیوندهای کوالان از انتهای ‘5 به قطعات DNA الگو متصل می شوند.
مرحله تکثیر در این روش به صورت bridge_PCR انجام می شود که در این مرحله از هر قطعه حدود یک میلیون نسخه دو رشته ای حاصل می شود که در نواحی کوچک به صورت خوشه های متمرکز قرار می گیرند . تکثیر شدت سیگنال مورد نیاز برای تشخیص در مرحله تعیین توالی را ضمانت می کند. پس از این مرحله، خوشه های واسرشت شده و به صورت تک رشته ای در می آیند که در مرحله بعد به کار می روند. به زبان ساده تر می توان گفت DNA تازه سنتز شده به منظور اتصال به آغازگر مجاور خم شده و به صورت پل در می آید و محصول تکثیر هر قطعه به شکل خوشه ای قرار می گیرد.
در مرحله بعدی که مرحله ی اصلی تعیین توالی است 4 نوع dNTP که هر یک دارای یک نوع رنگ فلورسنت می باشند توسط DNA پلیمراز به رشته ای که با کمک آنزیم Linearization تک رشته شده است افزوده می شوند. هر نوکلئوتید نشان دار که انتهای ‘3 آن مسدود است وقتی وارد می شود، واکنش برای مدتی متوقف می شود، نوکلئوتیدهای اضافی از محیط شکسته شده و به دو سمت قطعات، سازگاردهنده افزوده می شود.
سپس این قطعات به آغازگرهای متصل به سطح اسلاید flow cell متصل می شوند و تکثیر می گردند و سپس flow cell که سطحی مستحکم می باشد شستشو داده می شود و در این هنگام تصویر توسط دوربین ثبت می گردد و در نهایت مسدودیت انتهای ‘3 با حذف رنگ فلورسنت برطرف می شود . در واقع می توان گفت مرحله تعیین توالی روندی متوالی و تکرار شونده از ورود نوکلئوتید نشان دار، شستشو، ثبت تصویر، برش نشانه و ایجاد یک ‘3 آزاد است و در نهایت با آنالیز رنگ های ثبت شده تعیین توالی انجام می شود.
بر خلاف روش سنگر ، در این روش خاتمه زنجیره دائمی نیست و با برش رنگ فلورسنت ادامه واکنش از سر گرفته می شود به همین خاطر به این روش Cyclic reversible termination می گویند.
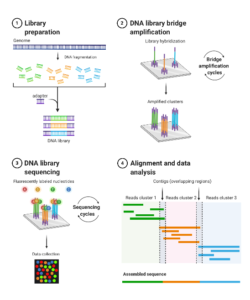
در مورد توالی یابی ایلومینا بیشتر بخوانید.
-
تعیین توالی یون تورنت(Semiconductor Sequencing/Ion Torrent)
این تکنیک متعلق به کمپانی Life Technologies بود که پس از مدتی کمپانی Thermo Fisher آن را خریداری کرد و دستگاه با نام Ion PGM ارائه شد. در این روش نیاز به اندازه گیری نور های فلوئورسانس نیست و مقدار H⁺ یا پروتون آزاد شده اندازه گیری می شود(Detecting of Hydrogen). از Emulsion PCR برای تکثیر قطعات استفاده میشود همچنین در این روش از یک سیستم شناسایی نیمه رسانا استفاده میشود.. در این روش هنگام قرار گیری یک نوکلئوتید در زنجیره ، یک پروتون آزاد می شود که منجر به ایجاد تغییر در PH می شود. این تغییر PH توسط سنسور بسیار حساس یونی شناسایی می شود.
چند سال بعد دستگاه پیشرفته تری با نام Ion proton وارد بازار شد که با حذف نیاز به استفاده از مواد فلورسنت و سنسور و دوربین، سرعت توالییابی افزایش و هزینههای آن کاهش پیدا کرد.
در مورد روش کار توالی یابی یون تورنت بیشتر بخوانید.
-
تعیین توالی SOLiD
تکنولوژی(Sequencing by Oligonucleotide Ligation and Detection) SOLiD از روش های نسل دوم توالی یابی بر اساس لیگیشن است که در سال 2006 ارائه گردید و در اواخر سال 2007 توسط شرکت ABI (Applied Bioscience) خریداری و تجاری سازی شد. از این فناوری برای توالییابی DNA استفاده میشود، اما به دلیل ماهیت موازی با توالی یابی نسل جدید، آنها در ترنسکریپتومیکس و اپی ژنومیکس نیز کاربرد دارند.
در روش توالی یابی SOLiD هر نوکلئوتید موجود در هر یک از قطعات 2 مرتبه، در 2 واکنش اتصال مستقل شناسایی می شود و در نتیجه مهم ترین مزیت این روش تعیین توالی میزان صحت %99.94 آن می باشد، از این رو، دقت توالی یابی در این روش در مقایسه با روش های دیگر در سطح بالایی است. همچنین سیستم این فناوری بر خلاف سیستم Roche 454 FLX توسط هموپلیمرها مانع نمی شود و بنابراین مناطق تکراری هموپلیمر بزرگ و دشوار دیگر برای توالی یابی مشکلی ندارند.
مهم ترین محدودیت این روش کوتاه بودن طول توالی خوانده شده می باشد که 35 تا 75 نوکلئوتید است. در ابتدا، در هر ران دستگاه تا 3GB توالی خوانده می شد، اما در دسامبر 2009، نسخه ی جدیدی از این تکنولوژی با نام Plus 3TM SOLiD معرفی گردید که می تواند در هر اجرا، بیش از 60GB توالی را بخواند.
در مورد روش توالی یابی SOLiD بیشتر بخوانید
نسل سوم توالی یابی
- SMRT
- Heliscope Single Molecular Seq
- nanopore DNA Seq
- Gnu Bio
-
تعیین توالی SMRT
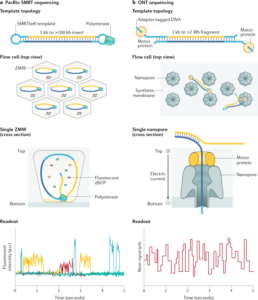
توالی یابی به روش SMRT یا single molecule real time را شرکت Pacific Bio sciences ابداع کرد. در این روش امکان مشاهده سنتز DNA در همان زمان وقوع میسر است. در سکوئنسینگ به روش SMRT ، آنزیم های DNA پلیمراز در ته چاهک ها (Zero Mode Waveguide) ثابت قرار می گیرند و نوکلئوتید های فلئوروسنت در سنتز شرکت می کنند و بر اساس فلئوروسنت آزاد شده ، توالی به دست می آید. متد توالی یابی کمپانی Pac Bio از جدید ترین روش های توالی یابی می باشد.
نرم افزارهای کاربردی برای آنالیز سکانس
FinchTV
Chromas
 آنالیز سکانس با نرم افزارهای FinchTV و Chromas رو به صورت خصوصی و ۶ ساعته یاد بگیرید!
آنالیز سکانس با نرم افزارهای FinchTV و Chromas رو به صورت خصوصی و ۶ ساعته یاد بگیرید!










































